Électricité statique : un exemple classique à l'école, dont on commence seulement à comprendre les mécanismes…
Le contexte, bref survol de ce qu'on sait déjà
En classe, on illustre souvent l'
électricité statique avec un ballon frotté sur les cheveux, Cf par exemple une activité
au 
.
Mais au delà de ces expériences assez bluffantes, les mécanismes de cette électrisation au niveau nanométrique sont mal compris.
On connaît d'étonnantes adaptations biologiques (des araignées dont le fil les maintient en l'air avec le champ électrique
JTS ci ; des bourdons munis de capteurs de champ électriques (Sutton,& al., 2016)
ici. On sait que ce phénomène jour un rôle la formation des planètes (Steinpilz,& al. (2020)
ici) et des nuages (Berdeklis,& al. 2001)
ici. De plus ce phénomène est au coeur de plusieurs applications industrielles (
imprimantes laser et photocopieuses, prévention des décharges électrostatiques, gestion des flux de poudres et poussières dans les conduites industrielles,
captation des particules fines des fumées de chauffage, etc.).
JTS présente ici une version vulgarisée : la
News dans Nature (Lacks, & al., 2025)
ici, et des extraits de l'article d'origine de Sobarzo et al. (2025) pour montrer que cela vaut la peine de s'y plonger pour tenter de bien comprendre…
 encourage le lecteur à aller vérifier dans l'article d'origine : ici
encourage le lecteur à aller vérifier dans l'article d'origine : ici Sobarzo et al. (2025) ici,(traduit) écrivent : "L'électrisation par contact, également appelée « tribo-chargement » ou « triboélectrification », défie notre compréhension. En principe, le phénomène semble simple : prenez deux isolants neutres, mettez-les en contact, puis séparez-les, et ils échangeront une charge électrique. Souvent associée à l'« électricité statique » et aux démonstrations classiques de ballons frottés sur les cheveux, l'électrisation par contact joue un rôle essentiel dans de nombreux phénomènes naturels, de l'électrification des nuages d'orage à l'adhésion du pollen sur les bourdons, en passant par l'accrétion de poussières dans les protoplanètes."  encourage le lecteur à aller vérifier dans l'article d'origine : ici (ils fournissent des références pour cest exemples)
encourage le lecteur à aller vérifier dans l'article d'origine : ici (ils fournissent des références pour cest exemples)
| La rationale d'un article scientifique est la justification logique qui motive l'étude. En quelques mots, c'est l'explication du pourquoi l'étude est menée, en s'appuyant sur l'importance du sujet et les lacunes de la littérature existante. |
La rationale = ce qu'on ne sait pas encore et que l'étude va aborder
Bien qu'on les ait empiriquement classés, on ignore pourquoi certains matériaux gagnent une charge négative et d'autres une charge positive. Lorsqu'un élève veut comprendre en profondeur ce qui se passe précisément pour que ces électrons se déplacent, on peut avancer un début d'explications, mais on est vite emprunté et en général on néglige des aspects plus complexes, comme le rôle des nano-déformations de surface dans ces transferts de charge.
D'après la news dans Nature Lacks, & al. (2025) ici Sobarzo et al. (2025) ici, sont plus explicites "Cependant, comme le rappellent systématiquement les articles sur le sujet, les aspects les plus fondamentaux du phénomène – à savoir la nature des porteurs de charge et les causes de leur transfert – restent débattus. Parmi les observations les plus marquantes figure la tendance des matériaux à s'ordonner en séries triboélectriques, c'est-à-dire en listes transitives basées sur le signe de la charge qu'ils acquièrent. Par exemple, dans la première série de ce type, établie par J. C. Wilcke en 1757, le verre se chargeait positivement par rapport au papier, et le papier positivement par rapport au soufre ; il en résultait que le verre se chargeait positivement par rapport au soufre.
L'existence des séries triboélectriques a conduit à l'hypothèse qu'un unique paramètre sous-jacent pourrait dominer le phénomène – appelons-le φ par convention. De nombreux mécanismes ont été proposés pour expliquer φ, parmi lesquels les propriétés électroniques, l'acidité et la basicité, le potentiel zêta, l'hydrophilie et l'hydrophobie, la flexoélectricité et la mécanochimie, pour n'en citer que quelques-uns." Traduction de Sobarzo et al. (2025)  encourage le lecteur à aller vérifier dans l'article d'origine : ici
encourage le lecteur à aller vérifier dans l'article d'origine : ici
Cette nouvelle recherche et ce qu'elle apporte
La
News (Lacks, & al., 2025)
ici présente le dispositif expérimental
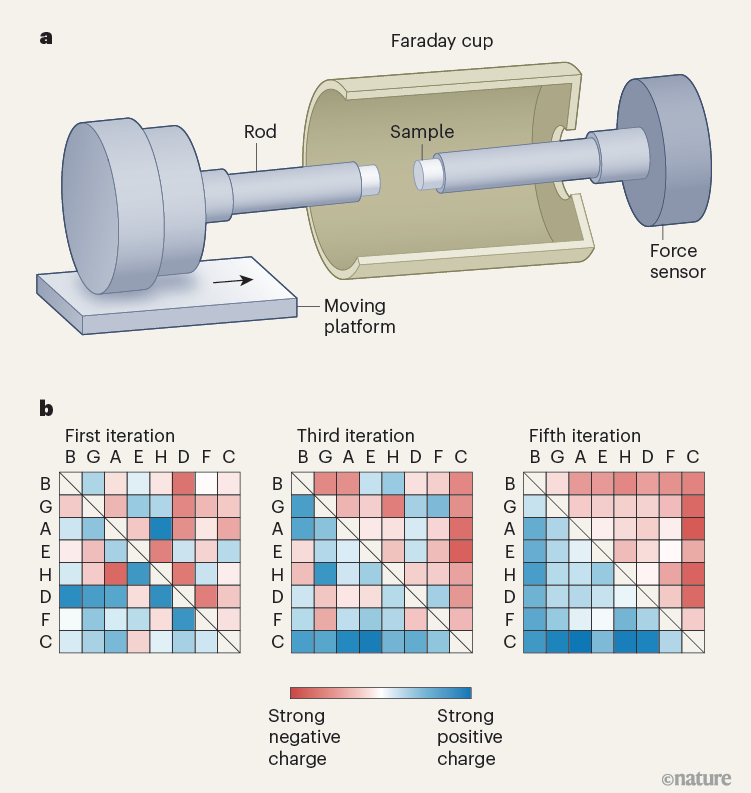
Fig 1: Les propriétés de transfert de charge de matériaux identiques divergent spontanément pour former une série distincte. a. Dans les expériences de Sobarzo et al., des échantillons identiques d'un matériau ont été fixés sur des tiges et pressés l'un contre l'autre, en utilisant un capteur de force pour garantir l'application d'une pression définie. L'échange de charge entre les échantillons a été mesuré à l'aide d'un dispositif capturant les particules chargées (une coupelle de Faraday) relié à un électromètre.. [figure]. Source : Sobarzo et al. (2025)
L'étude de Sobarzo et al. (2025) apporte un
début d'éclairage nouveau et assez surprenant : La news (Lacks, & al., 2025)
ici la résume en indiquant que des matériaux en contact « se souviennent » de leurs interactions passées.
D'après Nature Lacks, & al. (2025) ici
Franchement JTS a trouvé cela étrange, fascinant, presque mystique, mais pas très éclairant.
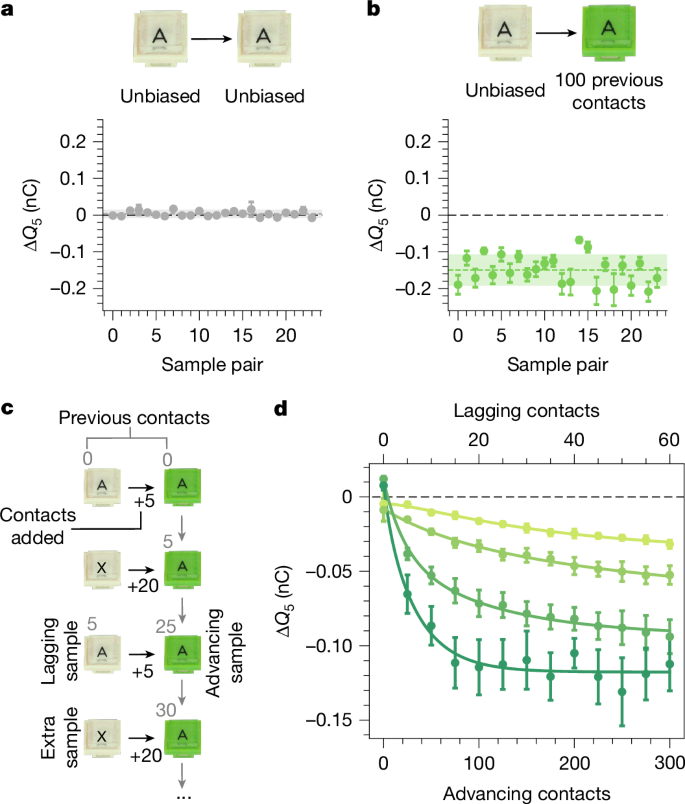
En lisant Sobarzo et al. (2025) ici, on trouve qu'en mesurant le ΔQ (charge moyenne échangée sur cinq contacts, en commençant toujours avec des échantillons entièrement déchargés (voir Methods).) alors qu'au premier frottement, ils sont à peine chargés et de polarité aléatoire (leur figure 2a), plus les échantillons ont subi de frottements, plus ils se chargent ensuite -et toujours négativement (fig 2 b) Traduction de Sobarzo et al. (2025)  encourage le lecteur à aller vérifier dans l'article d'origine : ici
encourage le lecteur à aller vérifier dans l'article d'origine : ici
Fig 2: Pour explorer les causes de l'évolution de la série, nous avons d'abord effectué des mesures de référence sur 24 paires d'échantillons vierges. Ces derniers se chargent aléatoirement autour de zéro, […]b. Motivés par l'hypothèse que l'acte de contact lui-même pourrait induire l'évolution de la série, nous avons préparé 48 nouveaux échantillons et exposé la moitié à 100 contacts préalables. En les mettant en contact avec des échantillons n'ayant pas subi de contacts précedemment (unbiased) et en mesurant ΔQ₅, nous avons observé un effet net : les échantillons ayant subi des contacts préalables se chargent systématiquement négativement. [figure]. Source :figure 2 Sobarzo et al. (2025)
Ainsi quand la news parle de "mémoire" il s'agit d'une accumulation ΔQ au cours des essais, JTS trouve qu'on comprend mieux en lisant l'article d'origine :-)
Un modèle : Premier paramètre proposé : l'historique des contacts (~leur intégrale)
Sobarzo et al. (2025) produisent ensuite un modèle qui reproduit leurs données expérimentales et les séries triboélectriques. Ils montrent que ce modèle explique ces données et permet de faire des prédictions qu'on peut vérifier
Voir le texte de Sobarzo, & al. (2025)
ici, et un extrait :
 encourage le lecteur à aller vérifier dans l'article d'origine : ici
encourage le lecteur à aller vérifier dans l'article d'origine : ici In this picture, the ordering of the triboelectric series at any instant should correspond to the ordering of the different potentials. However, it is clear from Fig. 2d that contact causes changes to the potentials, that is, dφi/dni = f(ni, φi,…), in which ni is the number of contacts sample i has experienced. The critical idea is now this: if contact causes the potentials to ultimately separate, then any ensemble will eventually result in a triboelectric series. Moreover, if the ordering of the final potentials, , is not the same as , it must be the case that, during their evolution, they 'crossed', resulting in what we (incorrectly) perceive as 'cycle
We can use this hypothesis to develop a numerical model that reproduces our experimental data.
Figure 2d implies an evolution consistent with the following differential equation,
in which
αi are growth rates and

are the values of the potentials at infinite contacts.
Ensuite ils valident ce modèle avec des données prédites et des séries triboélectriques cf. leur
figure 3.
 encourage le lecteur à aller vérifier dans l'article d'origine : ici
encourage le lecteur à aller vérifier dans l'article d'origine : ici
2ème paramètre proposé ; La mécanique du contact à l'échelle nanométrique
Alors qu'on n'avait pas observé d'effet clair des frottements dans d'autres études, Sobarzo, et al. (2025) étudient en détail ces interactions et trouvent un lissage aux fréquences spatiales élevées (c.-à-d. les plus "fines" irrégularités de surface), qui parait être lié à l'électrification de ces contacts.
Il est clair que le contact modifie fondamentalement la surfaces des échantillons d'une manière qui affecte le mécanisme d'électrification par contact. Ces altérations peuvent être élémentaires (par exemple, modification de la composition atomique), moléculaires (affectant les liaisons) ou physiques (par exemple, modification de la structure/morphologie). Dans la Fig. 4,, nous présentons un ensemble de tests variant la surface afin de détecter ces changements. À l'échelle atomique, d'autres expériences sur l'électrification par contact entre différents matériaux avaient montré que le contact entraîne des modifications de la composition élémentaire. […] Cependant, le calcul de la densité spectrale de puissance (PSD) des données AFM (atomic force microscopy) (Fig. 4h) révèle une caractéristique surprenante : les surfaces après contact sont plus lisses aux hautes fréquences spatiales. En réalisant de nombreux balayages en plusieurs endroits d'un même échantillon avant et après contact, nous constatons que ces différences sont statistiquement significatives. […] Nous montrons qu'en augmentant le nombre de contacts de 200 à plusieurs milliers, aucune modification n'apparaît dans les données LEIS, Raman et HD-SFG, mais l'adoucissement des queues de haute fréquence dans la PSD de rugosité s'accentue encore davantage. Ces observations suggèrent fortement que l'adoucissement des hautes fréquences présenté dans la Fig. 4h est bien la cause du biais de contact observé dans la Fig. 2 et, par conséquent, le moteur de l'auto-organisation spontanée dans notre système. (Traduction de Sobarzo, et al. (2025)
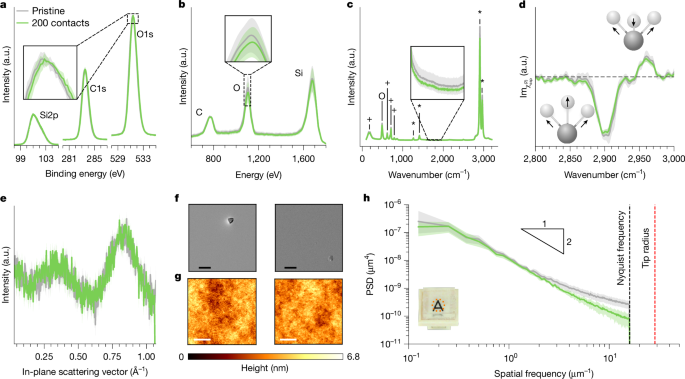
Fig 4: Ce qui change lors du contact 4h : Différences dans la Densité Spectrale de Puissance (PSD) de la rugosité, les échantillons ayant été en contact étant plus lisses aux hautes fréquences spatiales . Les bandes d'erreur représentent la dispersion d'environ dix mesures effectuées sur différentes régions d'un même échantillon dans ses états initial (pristine) et après contact, indiquant que ce résultat est statistiquement significatif. Légende et figre 4 complète voir ici [img]. Source :Sobarzo et al. (2025)
Une conclusion prudente
Sobarzo, & al. (2025) concluent qu'aborder le problème en termes de séries triboélectriques ne mène pas très loin, mais que l'étiudier en termes d'historique des contacts, et de mécanique du contact à l'échelle nanométrique serait une piste de recherche féconde.
Nous concluons que l'imprévisibilité de l'électrisation par contact n'est peut-être pas si insurmontable. En prêtant une attention rigoureuse à l'historique des contacts, nous pouvons non seulement expliquer cette imprévisibilité, mais aussi la maîtriser. En tenant compte de l'effet de l'historique des contacts, la notion de série triboélectrique peut être un outil heuristique utile, mais guère plus. Si cet effet est généralisé, chercher un ordre immuable reviendrait à poursuivre un mirage.
Enfin, en nous concentrant sur l'électrisation par contact entre matériaux identiques et sur des sources spécifiques d'imprévisibilité, nous isolons les éléments minimaux pertinents pour ses mécanismes. Face à un phénomène (1) aussi imprévisible et (2) étudié par des disciplines scientifiques distinctes, avec des langages et des cadres conceptuels différents, une telle clarification est précieuse. Dans notre système, elle nous permet de conclure que la cause du transfert de charge est intimement liée à la mécanique du contact à l'échelle nanométrique.Traduction de Sobarzo et al. (2025)  encourage le lecteur à aller vérifier dans l'article d'origine : ici
encourage le lecteur à aller vérifier dans l'article d'origine : ici
Les limites et les nouvelles questions qu'elle pose
Cette "mémoire" est évidemment assez troublante, et ne résout pas tout, mais stimule la curiosité des chercheurs, permet de nouvelles hypothèse et relance une question qui n'avançait guère.
Souvent, la science avance par petites touches dont on ne voit pas tout de suite en quoi elles seront importantes et mèneront à des applications (cf p. ex. le yaourt et la révolution CRISPR/Cas9 - JTS-ici). En lisant Sobarzo et al. (2025) ici, on trouve
"À la lumière de ces résultats, deux des mécanismes présentés dans l'introduction méritent une attention particulière : la mécanochimie et la flexoélectricité. Le premier suggère que la contrainte mécanique — qui devient extrêmement élevée lors de la déformation des aspérités à l'échelle nanométrique — peut suffire à provoquer une rupture hétérolytique des liaisons, entraînant ainsi la libération d'espèces chargées pour le transfert. Le second établit un lien entre la polarisation électrique et les gradients de contrainte mécanique — qui deviennent également importants lors de la déformation à l'échelle nanométrique — et peut donc être attendu pour produire d'importants champs électriques lors du contact.
Nos données ne suffisent pas à valider ou invalider directement l'une ou l'autre de ces hypothèses, mais elles nous incitent à une hypothèse intrigante : les deux pourraient être impliquées simultanément. Notre travail appelle ainsi à un examen plus approfondi de ces mécanismes, potentiellement en interaction, en mettant l'accent sur la nature fondamentalement tribologique de l'électrification par contact." Traduction de Sobarzo et al. (2025)  encourage le lecteur à aller vérifier dans l'article d'origine : ici
encourage le lecteur à aller vérifier dans l'article d'origine : ici
Cette recherche pourrait avoir des applications industrielles et même le problème des poussières sur la lune (Witze, et al. 2025)
ici, et sa toxicité (Pohlen et al., 2022)
ici .
Des perspectives pour l'enseignement
Ces résultats permettent d'aborder avec un angle nouveau des questions fondamentales en physique :
D'après la news dans Nature Lacks, & al. (2025) ici Quelles particules (électrons, ions, fragments de matière) sont impliquées dans l'électrisation ?
Pourquoi l'ordre de charge des matériaux (série triboélectrique) reste-t-il empirique et non prédictif ?
Sobarzo et al. (2025) apportent des réponses Comment des phénomènes à l'échelle nanométrique influencent-ils des effets macroscopiques observables en classe ?
Sobarzo et al. (2025) apportent des indices pour savoir où chercher Elle ouvre la voie à une meilleure compréhension des phénomènes électrostatiques - et ainsi d'ouvrir une de ces boites noires qu'on utilise souvent pour pudiquement cacher des explications complexes (Haskel-Ittah, 2023), Orange & Orange Ravachol, 2024)
"quelle boite noire peut-on / doit-on ouvrir ? Lesquelles doit-on laisser fermées, mais en évitant d'apparentes incohérences entre les faits retenus. S'il est impossible d'ouvrir, dans une étude donnée, toutes ces boites (dans le travail scientifique comme à l'école), les laisser toutes fermées pour éviter les problèmes et aller au plus vite à la « bonne solution » empêche toute problématisation approfondie. (Orange & Orange Ravachol, 2024 p. 101)
Peut-être saura-t-on bientôt ouvrir une boite noire de plus et expliquer ce qui se passe lorsqu'on retire un pull synthétique et qu'il attire les cheveux ?.
Remerciements
Remerciements à Laura Weiss pour une relecture détaillée et plusieurs commentaires constructifs qui ont amélioré cette publication.
- Berdeklis, P., & List, R. (2001). The Ice Crystal–Graupel Collision Charging Mechanism of Thunderstorm Electrification. Journal of the Atmospheric Sciences, 58(18), 2751‑2770. https://doi.org/10.1175/1520-0469(2001)058<2751:TICGCC>2.0.CO;2
- Haskel-Ittah, M. (2023). Explanatory black boxes and mechanistic reasoning. Journal of Research in Science Teaching, 60(4), 915‑933. https://doi.org/10.1002/tea.21817
- Lacks, D. J. (2012). The Unpredictability of Electrostatic Charging. Angewandte Chemie International Edition, 51(28), 6822‑6823. https://doi.org/10.1002/anie.201202896
- Lacks, D. J., & Sow, M. (2025). The secrets of static electricity are finally being revealed. Nature, 638(8051), 616‑618. https://doi.org/10.1038/d41586-025-00298-7
- Orange, C., & Orange Ravachol, D. (2024). Les « éducations à » sont-elles compatibles avec le développement d'une pensée critique en SVT ? Les didactiques face à l'évolution des curriculums. Savoir(s) et pratiques pour entrer dans la complexité du monde. Actes du 6ème colloque international de l'ARCD, 97‑109. https://archive-ouverte.unige.ch/unige:174755
- Pohlen, M., Carroll, D., Prisk, G. K., & Sawyer, A. J. (2022). Overview of lunar dust toxicity risk. Npj Microgravity, 8(1), 1‑9. https://doi.org/10.1038/s41526-022-00244-1
- Sobarzo, J. C., Pertl, F., Balazs, D. M., Costanzo, T., Sauer, M., Foelske, A., Ostermann, M., Pichler, C. M., Wang, Y., Nagata, Y., Bonn, M., & Waitukaitis, S. (2025). Spontaneous ordering of identical materials into a triboelectric series. Nature, 638(8051), 664‑669. https://doi.org/10.1038/s41586-024-08530-6
- Sutton, G. P., Clarke, D., Morley, E. L., & Robert, D. (2016). Mechanosensory hairs in bumblebees (Bombus terrestris) detect weak electric fields. Proceedings of the National Academy of Sciences, 113(26), 7261‑7265. https://doi.org/10.1073/pnas.1601624113
- Witze, A. (2025). Private spacecraft nails Moon landing : First images of Blue Ghost on the lunar surface. Nature. https://doi.org/10.1038/d41586-025-00670-7Steinpilz, T., Joeris, K., Jungmann, F., Wolf, D., Brendel, L., Teiser, J., Shinbrot, T., & Wurm, G. (2020). Electrical charging overcomes the bouncing barrier in planet formation. Nature Physics, 16(2), 225‑229. https://doi.org/10.1038/s41567-019-0728-9




![Scratching synergizes with FceRI mast cell activation to drive allergic skin inflammation. Crosslinking of FceRI or IgE on mast cells by allergens or S. aureus activates mast cell release of pruritogens (itch-inducing factors) that are sensed by MrgprA3-expressing neurons. Scratching resulting from itch sensation activates Trpv1-expressing neurons to release the neurotransmitter SP. SP acting through MrgprB2 on mast cells synergizes with FceRI to enhance mast cell release of TNF, resulting in enhanced cutaneous inflammation and increased S. aureus host defense. [Figure created with BioRender.com] Scratching synergizes with FceRI mast cell activation to drive allergic skin inflammation. Crosslinking of FceRI or IgE on mast cells by allergens or S. aureus activates mast cell release of pruritogens (itch-inducing factors) that are sensed by MrgprA3-expressing neurons. Scratching resulting from itch sensation activates Trpv1-expressing neurons to release the neurotransmitter SP. SP acting through MrgprB2 on mast cells synergizes with FceRI to enhance mast cell release of TNF, resulting in enhanced cutaneous inflammation and increased S. aureus host defense. [Figure created with BioRender.com]](https://www.science.org/cms/10.1126/science.adn9390/asset/069964a6-a3a2-45d0-8938-9e7ce53c3ec1/assets/images/large/science.adn9390-fa.jpg)