Comment le coronavirus infecte les cellules – et pourquoi Delta est si dangereux
Dans une news feature de Nature du 28 juillet, Megan Scudellari (ici) synthétise ce que les scientifiques sont en train de comprendre du cycle du SARS-CoV-2 (parfois partiellement francisé SRAS-CoV-2) et les mécanismes par lesquels le virus échappe trop souvent à la détection par le système immunitaire.
Habituellement Jump-To-Science incite
surtout à aller voir l'article d'origine, mais vu l'importance
de cette question, JTS vous propose ici un résumé et une
traduction de cette news feature.
Des exemples d'implications éducatives choisies pour leur
possible pertinence aux programmes et à leur révision en cours,
sont proposées en vert. Chacun se sentirart libre d'adapter,
modifier ou rejeter en fonction de ses pédagogies, de ses
élèves, etc.
Les scientifiques ont découvert des adaptations-clés qui aident
le virus à s'accrocher aux cellules humaines avec une force
surprenante, puis à se cacher une fois à l'intérieur. Plus tard,
lorsqu'il quitte les cellules, le SARS-CoV-2 exécute une étape de
traitement cruciale pour préparer ses particules à infecter encore
plus de cellules humaines. Ce sont quelques-uns des outils qui ont
permis au virus de se propager si rapidement et de faire des
millions de morts.
De nombreux chercheurs étudient le processus d'infection, afin de trouver de meilleurs moyens de l'interrompre grâce à des traitements et des vaccins améliorés. Ils veulent aussi comprendre pourquoi les dernières souches, telles que le variant Delta, sont plus transmissibles.
Pourquoi la protéine Spike n'est-elle pas bien détectée par le
système immunitaire ?
 Fig
1: séquence animée du virus SARS-CoV-2. Une simulation
informatique de la structure du coronavirus SARS-CoV-2. [img].
Source :Janet
Iwasa, Université de l'Utah
Fig
1: séquence animée du virus SARS-CoV-2. Une simulation
informatique de la structure du coronavirus SARS-CoV-2. [img].
Source :Janet
Iwasa, Université de l'Utah
Fig 2 : Structure de la protéine
Spike du SARS-CoV-2 et son revêtement de glycanes (orange) RBD
en vert . [img]. Source : Image structurelle de Lorenzo Casalino,
Univ. Californie, San Diego (Réf. 1) Graphique : Nik Spencer/Nature
On y voit une boucle qui n'est pas couverte de glycane et qui
dépasse du sommet de la protéine, c'est un des 3 domaines de
liaison (Receptor Binding Domain RBD) aux récepteurs des
cellules humaine.
encourage le lecteur à aller vérifier dans
l’article d’origine : ici
Le modèle numérique de la protéine révèle une piste pour bloquer
l'entrée du Virus ?
Une équipe bioinformatique conduite par Ronnie Amaro, (Casalino et al., 2020), a développé
un modèle qui permet la visualisation la plus détaillée à ce jour,
basée sur des données structurelles et génétiques et rendues atome
par atome par un superordinateur. Ce modèle a permis d'explorer la
dynamique de cette protéine spike : lorsque le RBD s'est élevé
au-dessus du nuage de glycane, deux glycanes ont plongé pour
verrouiller cette position "dégainée", ici comme un cran d'arrêt.
Quand l'équipe d'Amaro a muté les glycanes dans le modèle
informatique, le RBD affaissé ( et donc moins à mème de se fixer
sur nos cellules).
L'équipe de McLellan (Casalino et al., 2020) a mis au
point un moyen d'essayer la même expérience en laboratoire et, en
juin 2020, ils avaient signalé que la mutation des deux glycanes
réduisait la capacité de la protéine Spike à se lier à un
récepteur cellulaire humain – un rôle que personne n'avait
auparavant identifié dans les coronavirus, dit McLellan. Il est
possible que l'extraction de ces deux sucres réduise
l'infectiosité du virus, dit Amaro, bien que les chercheurs
n'aient pas encore le moyen de le faire.
encourage le lecteur à aller vérifier dans
l’article d’origine : ici
Quelques implications éducatives
Alors que la biologie numérique est - depuis la
fin du siècle passé - au coeur de l'activité des chercheurs en
biologie (Morange, 2003) et que l'école doit désormais
intégrer la biologie numérique, cet exemple illustre bien
comment s'articulent et se complètent la biologie classique
(moléculaire) et actuelle numérique.
On peut noter que la modélisation développée par
l'équipe d'Amaro ne sert pas seulement à illustrer mais
intègre les affinités et dynamiques des atomes entre eux, ce qui
permet aussi d'explorer les fonctionnements, de prédire et
expliquer, c'est-à-dire de comprendre au sens du
biologiste.
Ne serait-il pas judicieux de savoir utiliser ces
modèles pour aider les élèves à comprendre... ?
- La protéine Spike détaillant à différents niveaux de zoom les structures et leur dynamique (MP4)
- Gros plan sur la tete de Spike avec le RBD en position "dégainée" (MP4)
Ces vidéos illustrent bien la dynamique de
ces molécules et combien les interactions entre parties de la
protéine changent sa forme pour stabiliser en position active
(dégaînée) le RBD par exemple. Ce que les illustrations dans les
livres ne peuvent pas faire,... Sans parler de l'immense saut
des savoirs qui a été fait depuis leur publication …
ils offrent aussi
- 2 Structures 3D PDB de Spike " wild-type, open and closed full-length models of spike protein " (ZIP)
Imprimer un modèle de Spike que les élèves puissent manipuler pour mieux comprendre - On veillera à respecter les consignes sanitaires : désinfecter les objets par exemple.
Le projet Bioinformatique : opportunités pour l’enseignement indique comment ces structures pourront être visualisées, adaptées pour les imprimer en 3D. Notamment ces scénarios- Plus complet :Partir des donéees de Casalino,… & Amaro, (2020) Convertir la structure de la protéine en fichier pour imprimante 3D(.STL)
- Plus simple : trouver Spike dans cette table Nombreuses protéines avec leur structure et le fichier STL - prêtes pour impression
Le point sur ce qu'on sait du cycle du virus.
Scudellari (2021) synthétise les
derniers 19 mois de recherches dans le monde, appuyées sur des
décennies de recherche sur les (autres)
coronavirus, et décrit la façon dont le SARS-CoV-2 envahit les
cellules humaines. Cf. fig. 3.
Cycle de vie du coronavirus pandémique :
1 L’entrée dans la cellule commence avec la fixation des protéines Spike
Chaque virion (particule virale) du SARS-CoV-2 a une surface externe parsemée de 24 à 40 protéines Spike disposées au hasard qui sont la clé de la fusion avec les cellules humaines. Si pour d'autres types de virus, comme la grippe, les protéines de fusion externes sont relativement rigides, les protéines Spike du SARS-CoV-2 sont extrêmement flexibles et s'articulent en trois points.cf ici
Cela permet à Spike de basculer, de se balancer et de tourner, et semble leur permettre de scanner plus facilement la surface cellulaire et permettrait à plusieurs Spike de se lier à une cellule humaine. Il n'y a pas de données expérimentales similaires pour d'autres coronavirus, mais comme les séquences protéiques de Spike sont hautement conservées au cours de l'évolution, il raisonnalbe de supposer que le trait est partagé, dit Beck (Turoňová, 2020.Une
fois que les protéines virales Spike se lient à l'ACE2, d'autres
protéines à la surface de la cellule hôte initient un processus
qui conduit à la fusion des membranes virales et cellulaires (Cf.
fig. 6 et fig. 3 étape 1).
 Fig 6
: Entrée virale :
infographie montrant l'interaction entre les protéines Spike
et les récepteurs de l'hôte avant l'entrée virale. [img]. Source :
Janet Iwasa, Univ.
Utah; Graphique : Nik Spencer/Nature
Fig 6
: Entrée virale :
infographie montrant l'interaction entre les protéines Spike
et les récepteurs de l'hôte avant l'entrée virale. [img]. Source :
Janet Iwasa, Univ.
Utah; Graphique : Nik Spencer/NatureCf. fig 7 et fig. 3 étape 1.
 Fig 7 : Une animation de la fusion du SARS-CoV-2 avec
les cellules. [img]. Source :
Janet Iwasa, Univ. Utah;
Fig 7 : Une animation de la fusion du SARS-CoV-2 avec
les cellules. [img]. Source :
Janet Iwasa, Univ. Utah;
Implications éducatives
Se contenter d'une explication qui nous plait
quand on n'a pas pris la peine d'éliminer les autres
explications possibles n'est pas vraiment scientifique, mais
tellement humain !
Il faudrait tenter de réfuter sa propre croyance en examinant
soigneusement si c'est bien la seule conclusion possible…
Cette rigueur critique a été difficile pour de
nombreuses personnes qui ont repris cette étude sans en discuter
les limites parce qu'ils avaient tellement envie que tel
traitement soit efficace... et leur renommée sauvegardée.
C'est humain, socialement et politiquement compréhensible, mais
ce n'est pas la démarche scientifique que nous devrions
enseigner aux élèves.
une démarche qui les aide à se construire une opinion, à
dépasser l’argument d'autorité (XX est un spécialiste … plus
sérieux que le tien !).
On voit aussi l'importance d'examiner les
méthodes: le simple fait que cette étude utilisait des cellules
qui pouvaient différer de ce qui se passe dans le corps humain
(en clinique) n'a pas été pris en compte par ceux qui se sont
précipités sur la conclusion parce qu'elle convenait à leurs
croyances. Le nombre de participants et le fait qu'ils n'ont pas
été choisis au hasard permet de comprendre le scepticisme face à
certaines études (ici
p. ex)
"Chercher pourquoi on a raison est à la portée de
tous. La démarche scientifique consiste à chercher pourquoi on a
tort, et de ne conclure que l'on a raison que si l'on n'y arrive
pas.» Hurst, Samia (2020) ici
C'est si difficile à faire
avec les programmes surchargés… mais peut-être que les
révisions en cours sont une opportunité ?
La différence entre faire adhérer à ses idées et aider les élèves à utiliser la science pour construire leurs idées :
- Potvin, P. (2021), Enseigner pour convaincre—Former les élèves à l’esprit scientifique dans un monde qui en a bien besoin. Spectre. Consulté 23 août 2021, à l’adresse https://spectre.s1.yapla.com/fr/accueil/enseigner-pour-convaincre---former-les-eleves-a-l-esprit-scientifique-dans-un-monde-qui-en-a-bien-besoin
Pour approfondir vraiment ce qu'est la
démarche scientifique on peut recommander l’ouvrage de
Jean-YvesCariou : Histoire des démarches scientifiques - De
l’Antiquité au monde contemporain (2019) Editions Materiologiques. Ou des articles :
- Cariou, J.-Y. (2010). Les opinions
vulnérables, tremplin vers le savoir. RDST. Recherches en
didactique des sciences et des technologies, 1, 67‑92. https://doi.org/10.4000/rdst.163
Cariou, J.-Y. (2019). Discipliner l’esprit scientifique d’Apprenants-Stratèges. VOLET 1–Conférences en plénière du Colloque international du DIDACTIfen 2018, 37. ici
Réorientation du
fonctionnement de la cellule vers la production de virus
Les étapes suivantes de l'infection sont actuellement moins clairement comprises. « Il y a plus d’incertitude et d’hypothèses concurrentes. […] une fois que vous êtes à l'intérieur de la cellule", explique la chimiste Janet Iwasa de l'Université de l'Utah à Salt Lake City, qui développe une animation annotée du cycle de vie viral - notamment celles figurant dans cette publication (fig.1, 6, 7) .
Les Coronavirus modifient les fonctionnements de cette machinerie de plusieurs manières. (cf. figure 3 étape 3)
Une fois que le virus a introduit son ARN génomique dans la cellule, les ribosomes du cytoplasme humain traduisent deux sections d'ARN viral en longues chaînes d'acides aminés, qui sont ensuite découpées en 16 protéines, dont beaucoup sont impliquées dans la synthèse de l'ARN viral. Plus tard, davantage d'ARN sont générés qui codent pour un total de 26 protéines virales connues, y compris les protéines structurelles utilisées pour fabriquer de nouvelles particules virales, telles que Spike, et d'autres protéines accessoires. De cette façon, le virus commence à produire des copies de son propre ARN. Comme d'autres virus il a besoin de la machinerie de la cellule pour traduire ces ARNm en protéines (cf. figure 3 étape 3).
Suppression de
traces qui révèleraient le virus
La virologue Noam Stern-Ginossar et son équipe (Finkel, et al.,2021) ont zoomé sur trois mécanismes par lesquels le SARS-CoV-2 supprime la traduction de l'ARNm de l'hôte en faveur du sien. Aucun n'est exclusif à ce virus, mais la combinaison, la vitesse et l'ampleur des effets semblent uniques, explique la chercheure.
Fig 8 : Pourcentage des ARN de l’hôte (bleu) et
viraux (rouge) en fonction du temps (hpi=heures post infection).
[img]. Source : Figure 1 de Finkel, et
al.,2021
Premièrement, le virus élimine la compétition : la
protéine virale Nsp1, l'une des premières protéines traduites dès
l'entrée du virus, recrute des protéines hôtes pour hacher
systématiquement tous les ARNm cellulaires qui n'ont pas
d'étiquette virale.
Lorsque l'équipe de Stern-Ginossar a mis cette même étiquette à
l'extrémité d'un ARNm hôte, l'ARNm n'a pas été découpé.
Deuxièmement, l'infection réduit la traduction globale des protéines dans la cellule de 70 %. Nsp1 est à nouveau le principal coupable, bloquant cette fois physiquement le canal d'entrée des ribosomes afin que l'ARNm ne puisse pas pénétrer à l'intérieur, selon les travaux d'autres équipes de recherche. (Schubert, et al., 2020, Thoms, et al., 2020). La petite capacité de traduction qui reste est dédiée aux ARN viraux, explique Stern-Ginossar.
Enfin, le virus arrête le système d'alarme de la cellule. Cela se produit de nombreuses manières, mais l'équipe de Stern-Ginossar a identifié un mécanisme clair pour le SARS-CoV-2 : le virus empêche l'ARNm cellulaire de sortir du noyau, empêchjant la production des protéines destinées à alerter le système immunitaire en cas d'infection. Une deuxième équipe a confirmé ce résultat, et a de nouveau pointé du doigt Nsp1 : la protéine semble bloquer les canaux de sortie du noyau afin que rien ne puisse s'en échapper.
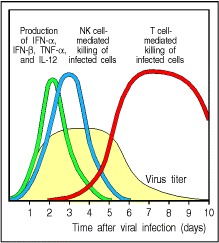 Parce que les transcrits génétiques ne peuvent
pas sortir du noyau, les cellules infectées ne libèrent pas beaucoup
d'interférons - ce sont des protéines de signalisation qui alertent
le système immunitaire de la présence d'un virus et font partie de
la réponse immunitaire inné (cf. Janeway fig
2.41 : IFN en vert dans la figure 9 ci-contre).
Parce que les transcrits génétiques ne peuvent
pas sortir du noyau, les cellules infectées ne libèrent pas beaucoup
d'interférons - ce sont des protéines de signalisation qui alertent
le système immunitaire de la présence d'un virus et font partie de
la réponse immunitaire inné (cf. Janeway fig
2.41 : IFN en vert dans la figure 9 ci-contre).Le SARS-Cov-2 est ainsi particulièrement efficace pour bloquer ce système d'alarme. Et en juin dernier, des chercheurs ont signalé des mutations dans le variant Alpha qui semblent lui permettre de freiner encore plus efficacement la production d'interféron.
"Il est clair que le SARS-CoV-2 est un virus très rapide qui a
une capacité unique à empêcher notre système immunitaire de
reconnaître et de combattre l'infection dans les premiers stades",
déclare Stern-Ginossar.
![]() encourage
le lecteur à aller vérifier dans l’article d’origine ici
encourage
le lecteur à aller vérifier dans l’article d’origine ici
Une réaction excessive qui peut être grave
Au moment où le système immunitaire réagit à la présence d'un
virus, ils son tellement abondants que les protéines de réponse
immunitaire inondent parfois la circulation sanguine à un rythme
plus rapide que la normale, ce qui peut causer des dommages. Les
médecins ont constaté au début de la pandémie que certaines
personnes très malades du COVID-19 sont lésées par une réponse
immunitaire hyperactive au SARS-CoV-2, en plus des effets du virus
lui-même. Voir notamment Wadman (2020) ici et JTS Comment
le COVID-19 attaque l'organisme
Certains traitements éprouvés à base de corticoïdes agissent ainsi paradoxalement sur le
formes graves en atténuant cette réponse
immunitaire.
La
cellule profondément transformée
Une
fois que le virus a pris le contrôle de la traduction de l'hôte,
ses gènes remodèle considérablement l'intérieur et l'extérieur de
la cellule.
Premièrement, certaines des protéines Spike virales nouvellement
fabriquées se déplacent à la surface de la cellule et sortent de
la membrane de la cellule hôte. Là, ils activent un canal ionique
à calcium présent dans la cellule hôte, qui expulse un revêtement
gras à l'extérieur de la cellule - le même revêtement que l'on
trouve sur les cellules qui fusionnent naturellement, telles que
les cellules musculaires. La cellule infectée fusionne alors avec
les cellules voisines exprimant l'ACE2, et se développent en
cellules respiratoires massives contenant jusqu'à 20 noyaux. Ces
structures fusionnées, appelées syncytia (pluriel
de syncitium pour les héllénistes),

Fig 10: Structures cellulaires fusionnées (syncytia)
observées dans les cellules exprimant la protéine Spike
SARS-CoV-2 (vert). Les noyaux sont en bleu et le squelette
cellulaire est en rouge. [img].Crédit : Mauro Giacca
Des syncitia permettent une reproduction durable du virus et la fusion avec des lymphocytes la dissémination ?
Le
biologiste moléculaire Mauro Giacca émet l'hypothèse que la
formation de syncytia permet aux cellules infectées de se
développer pendant de longues périodes, produisant de plus en plus
de virus durablement. (cf. figure 3 étape 3)
Une autre équipe, dirigée par Qiang Sun a découvert que certaines
cellules infectées par le COVID-19 forment même des syncytia avec
des lymphocytes (Zhang, et al., 2021). Il s'agit d'un
mécanisme connu d'évasion (qui permet
d'échapper…) immunitaire par les cellules tumorales,
mais pas par les virus. Cela suggère que les cellules infectées
évitent la détection immunitaire en s'accrochant simplement et en
fusionnant avec ces cellules immunitaires à proximité.
À l'intérieur de la cellule, encore plus de changements se
produisent. Comme d'autres coronavirus, le SARS-CoV-2 transforme
le réticulum endoplasmique (RE), un réseau de fines membranes
plates impliquées dans la synthèse et le transport des protéines,
en sphères à double membrane, comme si le RE soufflait des bulles.
Ces vésicules à double membrane (DMV) pourraient fournir un
endroit sûr pour la réplication et la traduction de l'ARN viral,
le protégeant des capteurs immunitaires innés dans la cellule,
mais cette hypothèse est toujours à l'étude.
![]() encourage
le lecteur à aller vérifier dans l’article d’origine ici
encourage
le lecteur à aller vérifier dans l’article d’origine ici
Comprendre
les mécanismes du virus permet d’explorer des pistes
thérapeutiques
Les
protéines impliquées dans la fabrication de ces vésicules à double
membrane (DMV) pourraient être de bonnes cibles médicamenteuses,
car elles semblent être indispensables à la réplication virale. La
protéine transmembranaire du coronavirus Nsp3 pourrait également
être une cible médicamenteuse potentielle : elle crée un pore en
forme de couronne dans les parois des DMV pour faire sortir l'ARN
viral 21 nouvellement fabriqué. La bloquer pourrait prévenir la
multiplication du virus. (cf. figure 3 étape 3)
La plupart des virus qui ont une enveloppe externe la forment en
s'assemblant directement au bord de la cellule, intégrant une
partie membrane plasmique de la cellule lors de sa sortie. Mais
les protéines de coronavirus nouvellement fabriquées empruntent un
chemin différent.
La
membrane formée à partir du complexe Golgi ou des lysosomes ?
Pendant
des années, des recherches ont suggéré que les coronavirus sont
transportés hors de la cellule via le complexe de Golgi, un
organite qui fonctionne comme un bureau de poste, emballant des
molécules dans des membranes et les envoyant vers d'autres parties
de la cellule. Là, le virus forme une enveloppe lipidique à partir
de la membrane du complexe de Golgi ; Les virions nouvellement
formés sont ensuite transportés à l'intérieur des vésicules de
Golgi jusqu'à la surface cellulaire, où ils sont éjectés hors de
la cellule, explique la virologue et biologiste cellulaire Carolyn
Machamer, qui a étudié les coronavirus pendant 30 ans.
Mais en décembre, la biologiste cellulaire Nihal Altan-Bonnet, et
ses collègues ont signalé qu'ils avaient détecté des coronavirus
quittant la cellule à avec des lysosomes – des vésicules
cellulaires pleines d'enzymes qui décomposent des fragments
cellulaires - parfois qualifiés de poubelles. Le blocage de la
voie sécrétoire basée sur Golgi n'a pas semblé affecter la
quantité de virus infectieux libéré, explique Altan-Bonnet. Les
preuves de son équipe suggèrent que les protéines virales forment
une enveloppe en bourgeonnant dans le RE, puis prennent le relais
des lysosomes pour sortir de la cellule. Les chercheurs testent
actuellement en tant que candidats antiviraux potentiels des
inhibiteurs qui bloqueraient le processus de sortie
lysosomale.(cf. figure 3 étape 4).
![]() encourage
le lecteur à aller vérifier dans l’article d’origine ici:
encourage
le lecteur à aller vérifier dans l’article d’origine ici:
La découpe finale ?
À la sortie de la cellule, une modification de plus rend ce virus infectieux : un clivage de cinq acides aminés prépare le virus à se lier à sa prochaine cible. (cf. figure 3 étape 4)
A la jonction des sous-unités S1 et S2 de Spike, le SARS-CoV-2 diffère des autres Coronavirus: « Parce que le site était inhabituel, nous nous sommes concentrés sur lui, et il s'est avéré qu'en effet ce site est essentiel pour l'invasion des cellules pulmonaires », explique Pöhlmann. Avec son équipe ils ont montré qu'une protéine de la cellule hôte appelée furine reconnaît et coupe ces 5 acides aminés – un clivage pour que le virus pénètre efficacement dans les cellules pulmonaires humaines (Hoffmann, et al. 2020).Ce n'est pas la première fois que des chercheurs identifient un site de clivage par la furine sur un virus ; les virus hautement pathogènes de la grippe aviaire en sont également porteurs et une étude aux Pays-Bas a également révélé que le coronavirus avec un site de clivage de la furine intact pénètre plus rapidement dans les cellules des voies respiratoires humaines que les autres (Mykytyn, et al., 2020).
La furine semble couper ce site pendant l'assemblage, ou juste avant la libération du virion. Ce timing pourrait expliquer pourquoi le virus sort par le Golgi ou les lysosomes, explique Tom Gallagher, virologue. «Le virus, une fois assemblé, se déplace dans un organite où il se trouve en présence de la protéase furine.»
Ce clivage entre les sous-unités S1 et S2, libère les protéines Spike - cela les amorce en somme - de sorte que lors de l'entrée dans la cellule suivante, la deuxième coupe par TMPRSS2, expose la zone hydrophobe qui s'enfouit alors rapidement dans la membrane de la cellule hôte. Sinon elles contournent TMPRSS2 et entrent par la voie endosomale plus lente, voire pas du tout. (cf. figure 3 étape 4)
La course aux médicaments antiviraux pour vaincre le COVID-19 – et la prochaine pandémie
On a observé que deux variants, Alpha et Delta, ont leurs sites de clivage furine mutés. Les deux changements rendent la séquence plus susceptible à la furine, ce qui signifierait plus de protéines Spike amorcées. D'autres coronavirus n'ont que 10% de Spike ainsi amorcées, mais chez SARS-CoV-2, ce pourcentage monte à 50 %, plus de 50 % pour le variant Alpha, et même 75% avec le variant Delta hautement transmissible,
Implications éducatives
On peut aider les élèves à saisir un implicite
dans l'approche sous-jacente de ces recherches : La maladie est un problème de protéines en trop,
en insuffisance ou défectueuse. Il n'est pas certain que les
élèves se représentent ainsi la maladie. Si au fond ils
perçoivent la maladie comme une
punition divine, une vengeance de la nature, ou un dérèglement, un
déséquilibre, la puissance explicative de ces recherches
pourrait avoir peu de sens. et la méfiance des traitements en
résultant partiellement expliquée.
Cf. ici
un des scénarios du Projet La biologie numérique :
quelles opportunités pour mieux enseigner ?
- Éprouver le rôle central des protéines dans le fonctionnement normal et la maladie -> Cf. page 6 dans le document produit à l'occasion d'une formation continue ici.pdf
Tout ce qu'on cherche encore à comprendre
La communauté scientifique n'effleure encore que la surface de la
compréhension du SARS-CoV-2. Les principales inconnues incluent le
nombre de récepteurs ACE2 nécessaires pour se lier à chaque protéine
Spike ; quand exactement le site S2 est clivé par TMPRSS2 ; et le
nombre de Spikes nécessaires pour la fusion virus-membrane
cellulaire. En avril 2020, une équipe de l'Université de Californie
à San Francisco a identifié au moins 332 interactions entre le
SARS-CoV-2 et les protéines humaines25.Il n'est pas facile de suivre le rythme des mutation du virus. Heureusement, conviennent les experts, jusqu'à présent la plupart des mutations observées sont associées à l'efficacité de la propagation du virus, et non à l'ampleur des dommages causés par le virus à l'hôte.
En juillet, une étude [Preprint].(Li, et al., 2021) a rapporté que le variant Delta s'est développé - par rapport aux versions antérieures du virus - plus rapidement (détectable en 4 jours plutôt que 6) et à des niveaux plus élevés (1000x plus !) dans les poumons et la gorge des personnes atteintes.
Mais on ne sait pas encore avec certitude comment les mutations de Delta ont boosté le variant de cette manière, explique Stern-Ginossar. "C'est quelque chose que de nombreux laboratoires essaient de comprendre."
![]() encourage
le lecteur à aller vérifier dans l’article d’origine ICI:
encourage
le lecteur à aller vérifier dans l’article d’origine ICI:
Voir aussi dans Nature
Comment un variant de coronavirus fréquente émousse nos défenses immunitaires
The race for antiviral drugs to beat COVID — and the next pandemic
Références :
- Cariou, J.-Y. (2019). Histoire des démarches scientifiques :
De l’Antiquité au monde contemporain. Éditions
matériologiques.
- Cariou, J.-Y. (2010). Les opinions vulnérables, tremplin
vers le savoir. RDST. Recherches en didactique des sciences et
des technologies, 1, 67‑92. https://doi.org/10.4000/rdst.163
Cariou, J.-Y. (2019). Discipliner l’esprit scientifique d’Apprenants-Stratèges. VOLET 1–Conférences en plénière du Colloque international du DIDACTIfen 2018, 37. ici - Casalino, L., Gaieb, Z., Goldsmith, J. A., Hjorth, C. K., Dommer, A. C., Harbison, A. M., Fogarty, C. A., Barros, E. P., Taylor, B. C., McLellan, J. S., Fadda, E., & Amaro, R. E. (2020). Beyond Shielding : The Roles of Glycans in the SARS-CoV-2 Spike Protein. ACS Central Science, 6(10), 1722‑1734. https://doi.org/10.1021/acscentsci.0c01056
- Finkel, Y., Gluck, A., Nachshon, A., Winkler, R., Fisher,
T., Rozman, B., Mizrahi, O., Lubelsky, Y., Zuckerman, B.,
Slobodin, B., Yahalom-Ronen, Y., Tamir, H., Ulitsky, I.,
Israely, T., Paran, N., Schwartz, M., & Stern-Ginossar, N.
(2021). SARS-CoV-2 uses a multipronged strategy to impede host
protein synthesis. Nature, 594(7862), 240‑245. https://doi.org/10.1038/s41586-021-03610-3
- Ghosh, S., Dellibovi-Ragheb, T. A., Kerviel, A., Pak, E.,
Qiu, Q., Fisher, M., Takvorian, P. M., Bleck, C., Hsu, V. W.,
Fehr, A. R., Perlman, S., Achar, S. R., Straus, M. R.,
Whittaker, G. R., Haan, C. A. M. de, Kehrl, J., Altan-Bonnet,
G., & Altan-Bonnet, N. (2020). β-Coronaviruses Use
Lysosomes for Egress Instead of the Biosynthetic Secretory
Pathway. Cell, 183(6), 1520-1535.e14. https://doi.org/10.1016/j.cell.2020.10.039
- Hoffmann, M., Kleine-Weber, H., & Pöhlmann, S. (2020). A
Multibasic Cleavage Site in the Spike Protein of SARS-CoV-2 Is
Essential for Infection of Human Lung Cells. Molecular Cell,
78(4), 779-784.e5. https://doi.org/10.1016/j.molcel.2020.04.022
-
Hurst, Samia (2020) Lu dans la presse, journal de l'UNIGE. (2020, mars 26). https://www.unige.ch/lejournal/ejournal/ejournal-08/presse08/
- Janeway, C. A., Travers, P., Walport, M., & Shlomchik,
M. (2001). Immunobiology. New York and London: Garland http://www.ncbi.nlm.nih.gov/books/NBK10757/
- Ke, Z., Oton, J., Qu, K., Cortese, M., Zila, V., McKeane, L., Nakane, T., Zivanov, J., Neufeldt, C. J., Cerikan, B., Lu, J. M., Peukes, J., Xiong, X., Kräusslich, H.-G., Scheres, S. H. W., Bartenschlager, R., & Briggs, J. A. G. (2020). Structures and distributions of SARS-CoV-2 spike proteins on intact virions. Nature, 588(7838), 498‑502. https://doi.org/10.1038/s41586-020-2665-2
- Li, B., Deng, A., Li, K., Hu, Y., Li, Z., Xiong, Q., Liu,
Z., Guo, Q., Zou, L., Zhang, H., Zhang, M., Ouyang, F., Su,
J., Su, W., Xu, J., Lin, H., Sun, J., Peng, J., Jiang, H., …
Lu, J. (2021). Viral infection and transmission in a large,
well-traced outbreak caused by the SARS-CoV-2 Delta variant
[Preprint]. Epidemiology. https://doi.org/10.1101/2021.07.07.21260122
- Morange, M. (2003). Histoire de la biologie moléculaire
(2ème edition 2003). La Découverte.
- Mykytyn, A. Z., Breugem, T. I., Riesebosch, S., Schipper,
D., van den Doel, P. B., Rottier, R. J., Lamers, M. M., &
Haagmans, B. L. (2021). SARS-CoV-2 entry into human airway
organoids is serine protease-mediated and facilitated by the
multibasic cleavage site. ELife, 10, e64508. https://doi.org/10.7554/eLife.64508
- Peacock, T. P., Goldhill, D. H., Zhou, J., Baillon, L.,
Frise, R., Swann, O. C., Kugathasan, R., Penn, R., Brown, J.
C., Sanchez-David, R. Y., Braga, L., Williamson, M. K.,
Hassard, J. A., Staller, E., Hanley, B., Osborn, M., Giacca,
M., Davidson, A. D., Matthews, D. A., & Barclay, W. S.
(2021). The furin cleavage site in the SARS-CoV-2 spike
protein is required for transmission in ferrets. Nature
Microbiology, 6(7), 899‑909. https://doi.org/10.1038/s41564-021-00908-w
- Potvin, P. (2021), Enseigner pour convaincre—Former les élèves à l’esprit scientifique dans un monde qui en a bien besoin. Spectre. Consulté 23 août 2021, à l’adresse https://spectre.s1.yapla.com/fr/accueil/enseigner-pour-convaincre---former-les-eleves-a-l-esprit-scientifique-dans-un-monde-qui-en-a-bien-besoin
- Reardon, S. (2021). How the Delta variant achieves its ultrafast spread. Nature. https://doi.org/10.1038/d41586-021-01986-w
- Schubert, K., Karousis, E. D., Jomaa, A., Scaiola, A., Echeverria, B., Gurzeler, L.-A., Leibundgut, M., Thiel, V., Mühlemann, O., & Ban, N. (2020). SARS-CoV-2 Nsp1 binds the ribosomal mRNA channel to inhibit translation. Nature Structural & Molecular Biology, 27(10), 959‑966. https://doi.org/10.1038/s41594-020-0511-8
- Scudellari, M. (2021). How the coronavirus infects cells—And why Delta is so dangerous. Nature, 595(7869), 640‑644. https://doi.org/10.1038/d41586-021-02039-y
- Thoms, M., Buschauer, R., Ameismeier, M., Koepke, L., Denk,
T., Hirschenberger, M., Kratzat, H., Hayn, M., Mackens-Kiani,
T., Cheng, J., Straub, J. H., Stürzel, C. M., Fröhlich, T.,
Berninghausen, O., Becker, T., Kirchhoff, F., Sparrer, K. M.
J., & Beckmann, R. (2020). Structural basis for
translational shutdown and immune evasion by the Nsp1 protein
of SARS-CoV-2. Science, 369(6508), 1249‑1255. https://doi.org/10.1126/science.abc8665
- Turoňová, B., Sikora, M., Schürmann, C., Hagen, W. J. H., Welsch, S., Blanc, F. E. C., von Bülow, S., Gecht, M., Bagola, K., Hörner, C., van Zandbergen, G., Landry, J., de Azevedo, N. T. D., Mosalaganti, S., Schwarz, A., Covino, R., Mühlebach, M. D., Hummer, G., Krijnse Locker, J., & Beck, M. (2020). In situ structural analysis of SARS-CoV-2 spike reveals flexibility mediated by three hinges. Science, 370(6513), 203‑208. https://doi.org/10.1126/science.abd5223
- Wadman, M., Couzin-Frankel, J., Kaiser, J., & Matacic, C. (2020, avril 17). How does coronavirus kill? Clinicians trace a ferocious rampage through the body, from brain to toes. Science | AAAS. https://www.sciencemag.org/news/2020/04/how-does-coronavirus-kill-clinicians-trace-ferocious-rampage-through-body-brain-toes
- Zhang, Z., Zheng, Y., Niu, Z., Zhang, B., Wang, C., Yao, X., Peng, H., Franca, D. N., Wang, Y., Zhu, Y., Su, Y., Tang, M., Jiang, X., Ren, H., He, M., Wang, Y., Gao, L., Zhao, P., Shi, H., … Sun, Q. (2021). SARS-CoV-2 spike protein dictates syncytium-mediated lymphocyte elimination. Cell Death & Differentiation, 1‑13. https://doi.org/10.1038/s41418-021-00782-





Aucun commentaire:
Enregistrer un commentaire