L'épigénétique : un mécanisme scientifique, ou un terme qu'on évoque pour soutenir sa théorie fétiche voire réfuter un "déterminisme biologique"... ?
Etienne Danchin (2021) évoque l'excès de popularité d'un concept souvent mal compris "Ce qui était auparavant incompris était attribué à des dieux, alors qu'aujourd'hui «c'est épigénétique »" Danchin, E. (2021) p. 63
Il est parfois évoqué pour soutenir la théorie fétiche d'un.e journaliste :
- "Et si l'épigénétique était la clé pour retrouver un équilibre parfait ?" Verley, F. (2021, juin 16). Comment influer sur nos gènes pour vivre plus longtemps et en meilleure santé ? Vogue
Ce terme est beaucoup évoqué en sciences sociales pour réfuter un "déterminisme biologique" mais sans toujours justifier, il me semble, de manière scientifique - en s'appuyant sur des résultats discutés à la lumière des méthodes.
D'autres l'évoquent plus rigoureusement, comme Dupras, C. (2017) pour remettre en question le cadrage conceptuel en termes de nature et culture (perçus comme séparés). Il met en évidence combien cette séparation nature-culture est floue et ancrée dans des questions politiques, éthiques très délicates.
D'autres, comme l'historien des sciences Michel Morange (un biologiste au départ) s'élève aussi contre cette opposition dans une vidéo
- Liberté épigénétique vs. déterminisme génétique : un mauvais procès Youtube
JTS se concentrera ici sur les aspects biologiques : présenter les mécanismes et quelques exemples bien étayés en termes scientifiques.
Une définition
"On peut définir l'épigénétique comme l'étude de tous les changements d'expression des gènes non dus à une variation dans la séquence nucléotidique de l'ADN et qui néanmoins sont transmis soit entre générations de cellules, soit entre générations d'organismes". Danchin, E. (2021, p. 64)
Cette définition très large s'applique à de nombreux mécanismes possibles ou bien établis.
Quels mécanismes ?
"Le mécanisme épigénétique le mieux documenté concerne des modifications chimiques sur les nucléotides de l'ADN. La plus connue d'entre elles implique l'ajout d'un radical méthyle, sur certaines des cytosines […] Chez les mammifères, plus un promoteur est méthylé, plus cela rend difficile la fixation de la [machinerie machinerie moléculaire de transcription]. Si de nombreuses cytosines ont un radical méthyle, la transcription ne se produit plus et le gène en question est silencé [on dit que les gènes non exprimés sont «silencés »] . La méthylation-déméthylation des cytosines constitue donc un puissant mécanisme d'expression différentielle des gènes. Danchin, E. (2021) p.65
Méthylation de l'ADN
Dans la mesure ou la compaction de la chromatine empêche la transcription (on utilise souvent l'anglicisme silencer) , des modifications qui décompactent l'ADN rendent plus accessibles les gènes qui sont donc plus exprimés.
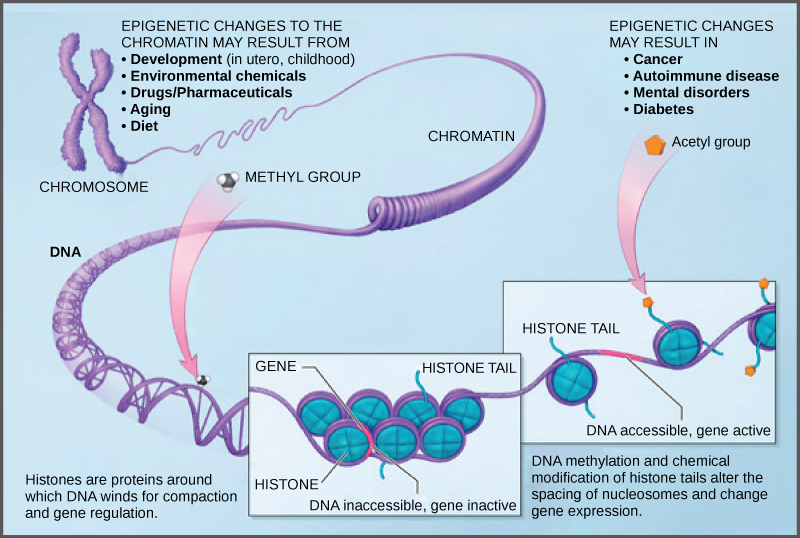
Fig 1: Commentaire img Source : WikimediaCNX Biology Textbook CC-BY-4
Modification des Histones
L'acétylation des histones peut aussi augmenter l'expression en favorisant l'accessibilité de l'ADN : cf. (Campbell & Reece,2015) p. 423
Il y a d'autres mécanismes comme l'ARN interférent. Voir notamment
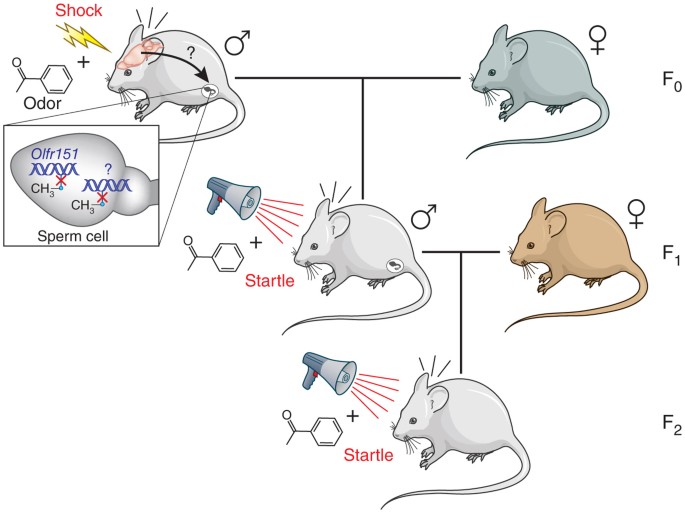
Un exemple bien documenté de mécanisme épigénétique chez la souris
Un exemple désormais classique trouvé dans la revue Nature et joliment intitulé " Lamarck revisited : Epigenetic inheritance of ancestral odor fear conditioning" Szyf, M. et al. (2014) ici
Fig 3 : Le conditionnement à craindre une odeur peut se transmettre à la génération suivante par modification épigénétique img source : Wikimedia CNX Biology Textbook CC-BY-4
- Szyf, M. (2014). Lamarck revisited : Epigenetic inheritance of ancestral odor fear conditioning. Nature Neuroscience, 17(1), 2‑4. https://doi.org/10.1038/nn.3603
Comment l'un des Chromosomes X est inactivé chez les femmes
Les mammifères femelles ont deux chromosomes X, et les hommes un seul. Un mécanisme remarquable a évolué qui empêche que ne se produise un déséquilibre dans l'expression de gènes : dans toutes les cellules en comportant deux, un chromosome X entier est «silencieux» c'est-à-dire que la transcription de l'ARN est empêchée. Ce processus s'appelle l'Inactivation du Chromosome X (XCI en anglais) et commence tôt dans le développement des embryons femelles. Une fois achevée cette inactivation XCI est essentiellement stable pour la vie. Par conséquence les gènes d'un chromosome X humain peut être réduit au silence pendant plus de 100 ans. Ce XCI est devenu un paradigme pour les processus épigénétiques - dans lesquels l'ADN et les protéines associées sont modifiés pour altérer l'expression des gènes. XCI a été étudiés de manière intensive pendant des décennies. Au cours des 25 dernières années, une grande partie de cette recherche s'est centrée sur un ARN long non codant (LNCRNA) appelée Xist, qui est nécessaire pour orchestrer cette XCI. Cependant, les détails du mécanisme d'inactivation (silencing en anglais) de Xist étaient restés incompris. Dossin et al. (2020) publient dans Nature une magnifique série d'expériences qui révèlent la manière dont les gènes Xist, en partenariat avec une protéine appelée Spen inactivent l'ADN de manière durable. (Traduction de Trotman, & al., 2020)  encourage le lecteur à approfondir dans l'article d'origine : ici
encourage le lecteur à approfondir dans l'article d'origine : ici

Fig 4: Mécanisme d'inactivation des gènes du X : l'ARN long non codant Xist et son cofacteur protéique, SPEN. répriment l'expression(silence en anglais) des gènes sur l'un des chromosomes X chez les cellules de mammifères femelles [img]. Source :Trotman, & al. (2020)
- Trotman, J. B., & Calabrese, J. M. (2020). How to silence an X chromosome. Nature, 578(7795), 365‑366. https://doi.org/10.1038/d41586-020-00207-0
- Dossin, F., Pinheiro, I., Żylicz, J.J. et al. SPEN integrates transcriptional and epigenetic control of X-inactivation. Nature 578, 455–460 (2020). https://doi.org/10.1038/s41586-020-1974-9
Comment le tabac modifie l'expression du Génome
- La protéine AHR dont l'expression est modifiée : P35869 @ Uniprot "Facteur de transcription activé par un ligand qui permet aux cellules de s'adapter aux conditions changeantes en détectant les composés de l'environnement, de l'alimentation, du microbiome et du métabolisme cellulaire, et qui joue un rôle important dans le développement, l'immunité et le cancer "
Comment des modifications épigénétiques permettent aux péruviens de vivre avec si peu d'oxygène
Les Quechua, un peuple autochtone du Pérou qui vit à des altitudes supérieures à 2500 mètres depuis au moins 11 000 ans, ont développé des adaptations héréditaires pour se perpétuer dans leur environnement hostile. Les scientifiques se demandent depuis longtemps si le stress environnemental, comme les faibles concentrations d'oxygène, peut façonner l'activité des gènes chez les peuples des hautes terres.[…] Cette recherche "pionnière" montre qu'il est non seulement possible, mais probable que des changements épigénétiques soient impliqués. Une nouvelle étude (Childebayeva, et al. 2021) ici révèle que dans ces conditions extrêmes on trouve des altérations épigénétiques […] de cet ADN, qui activent ou inactivent, diminuent ou augmentent l'activité de certains gènes. Ce mécanisme permet de s'adapter à l'environnement beaucoup plus rapidement que ne le permet la génétique classique. Le rôle que joue l'épigénétique pour aider les gens à s'adapter à une vie à haute altitude n'était pas clair avant cette étude. Cette recherche apporte les premières données expérimentales (evidence en anglais ~éléments de preuve) que grandir dans les montagnes peut altérer non seulement les gènes, mais aussi la façon dont ils sont exprimés dans le corps. (Traduction de (Kreier, F. 2020))  encourage le lecteur à approfondir dans l'article d'origine : ici
encourage le lecteur à approfondir dans l'article d'origine : ici
- Kreier, Freda (2020) High-altitude living has changed more than just the genes of some Peruvians. https://doi.org/10.1126/science.abg2903
- Childebayeva, A., Goodrich, J. M., Leon-Velarde, F., Rivera-Chira, M., Kiyamu, M., Brutsaert, T. D., Dolinoy, D. C., & Bigham, A. W. (2021). Genome-Wide Epigenetic Signatures of Adaptive Developmental Plasticity in the Andes. Genome Biology and Evolution, 13(2). https://doi.org/10.1093/gbe/evaa239
Une signature épigénétique unique propre aux jumeaux monozygotiques
Des chercheurs ont découvert que le jumeaux portent une signature épigénétique spécifique sur leur ADN, une signature qui se fixe dans leurs cellules au début du développement et se maintient jusqu'à l'âge adulte. À l'aide d'échantillons de sang et de cellules de la muqueuse buccale, les chercheurs ont scanné les épigénomes de plus plus de 3000 jumeaux monozygotiques, ainsi qu'un nombre comparable de jumeaux fraternels et de certains parents de jumeaux. Ils ont examiné 400 000 endroits différents sur le génome de chaque personne. Environ 800 sites présentaient des différences de méthylation qui distinguaient les jumeaux monozygotiques des autres, rapportent van Dongen, J.,et al. (2021). "C'est probablement quelque chose d'établi très tôt qui se propage aux cellules suivantes", explique van Dongen.
Un test épigénétique pourrait ainsi être utile pour déterminer si une personne a déjà eu un jumeau identique qui aurait disparu dans l'utérus. Parfois, un fœtus jumeau apparaît dans une échographie avant de disparaître, mais d'autres fois, il peut être absorbé sans laisser de trace. Selon certaines estimations, jusqu'à 12% des grossesses commencent comme multiples (y compris des jumeaux fraternels), mais seulement 2% des paires de jumeaux survivent. Traduction de Kayser, J. (2021).  encourage le lecteur à approfondir dans l'article d'origine : ici
encourage le lecteur à approfondir dans l'article d'origine : ici
- Kayser, J. (2021). Identical twins carry genetic modifications no one else has [Data set]. https://doi.org/10.1126/science.acx9209
- van Dongen, J., Gordon, S. D., McRae, A. F., Odintsova, V. V., Mbarek, H., Breeze, C. E., Sugden, K., Lundgren, S., Castillo-Fernandez, J. E., Hannon, E., Moffitt, T. E., Hagenbeek, F. A., van Beijsterveldt, C. E. M., Jan Hottenga, J., Tsai, P.-C., Min, J. L., Hemani, G., Ehli, E. A., Paul, F., … Boomsma, D. I. (2021). Identical twins carry a persistent epigenetic signature of early genome programming. Nature Communications, 12(1), 5618. https://doi.org/10.1038/s41467-021-25583-7
Le placenta : rôle-clé de modifications épigénétiques
Plus tôt ce mois-ci dans Nature, Li, et al. (2020), ont rapporté avoir trouvé de l'ADN méthylée (méthyladénine) dans des cellules des cellules souches du trophoblaste (qui deviendra placenta) de souris. Les méthyladénines étaient fréquentes dans les régions actives du génome, où la double hélice de l'ADN se déroule brièvement. Li et al. ont découvert que les méthyladénines dans l'ADN déroulé empêchent la liaison de la protéine SATB1 nécessaire pour rembobiner l'ADN. Le déroulement bloque à son tour l'expression des gènes qui permettraient aux cellules souches du trophoblaste de se différencier et d'arrêter leur croissance. Au lieu de cela, les cellules se multiplient, assurant qu'il y en a suffisamment pour former le placenta complet. Plus tard, les méthyles sont retirés - les chercheurs ne savent pas comment - et les cellules commencent à se spécialiser pour fabriquer le placenta. Traduction de Pennisi, E. (2021).  encourage le lecteur à approfondir dans l'article d'origine : ici
encourage le lecteur à approfondir dans l'article d'origine : ici
Fig 7: Chez la souris, l'adénine méthylée stabilise l'ADN déroulé en empêchant la liaison par la protéine SATB1. La méthyladénine permet aux cellules souches de se multiplier avant de se différencier pour former le placenta. . [img]. Source :V. Altounian/Science in Pennisi, E. (2021)
- Pennisi, E. (2021). Controversial DNA modification could play key role in placenta development. https://doi.org/10.1126/science.abd9820
- Li, Z., Zhao, S., Nelakanti, R. V., Lin, K., Wu, T. P., Alderman, M. H., Guo, C., Wang, P., Zhang, M., Min, W., Jiang, Z., Wang, Y., Li, H., & Xiao, A. Z. (2020). N6-methyladenine in DNA antagonizes SATB1 in early development. Nature, 583(7817), 625‑630. https://doi.org/10.1038/s41586-020-2500-9
L'age du chien établi par les modifications épigénétiques ?
Cette étude est basée sur un concept relativement nouveau dans la recherche sur le vieillissement : que les modifications chimiques de l'ADN d'une personne au cours d'une vie créent ce qu'on appelle une horloge épigénétique. Les scientifiques ont démontré qu'une de ces modifications, l'ajout de groupes méthyle à des séquences d'ADN spécifiques, suit l'âge biologique de l'homme, c'est-à-dire les effets que la maladie, un mode de vie malsain et la génétique font subir à notre corps. En conséquence, certains chercheurs ont converti le statut de méthylation de l'ADN d'une personne en une estimation de l'âge - ou même une prédiction de l'espérance de vie (des éthiciens inquiets, disent que les données pourraient être utilisées à mauvais escient par les enquêteurs médico-légaux et les compagnies d'assurance (Dupras, C., et al. (2019) ici) .
D'autres espèces subissent également une méthylation de l'ADN en vieillissant. Les souris, les chimpanzés, les loups et les chiens, par exemple, semblent tous avoir des horloges épigénétiques. Pour découvrir en quoi ces horloges diffèrent de la version humaine, le généticien Trey Ideker de l'Université de Californie à San Diego et ses collègues (Wang, et al.… Ideker, 2019) ici)ont commencé avec des chiens. Même si les meilleurs amis de l'homme ont divergé des humains au début de la divergence des mammifères, ils constituent un bon groupe de comparaison car ils vivent dans les mêmes environnements et beaucoup reçoivent des soins de santé et des traitements hospitaliers similaires.  encourage le lecteur à approfondir dans l'article d'origine : ici
encourage le lecteur à approfondir dans l'article d'origine : ici
Dans l'ensemble, l'horloge épigentique canine tourne beaucoup plus vite au départ que celle de l'humain - un labrador de 2 ans peut se comporter comme un chiot mais il est d'âge moyen, suggère ce modèle basé sur la méthylation, puis ralentit.
La nouvelle formule, qui s'applique aux Labradors estime l'âge humain d'un chien à peu près :
âge équivalent humain = 16 x ln(âge du chien) + 31
C'est-à-dire le logarithme naturel (ln) de l'âge réel du chien, multiplié par 16, avec augmenté de 31
- Morell, V. (2019). Here's a better way to convert dog years to human years, scientists say. Science. https://doi.org/10.1126/science.aba2340
- Dupras, C., Beck, S., Rothstein, M. A., Berner, A., Saulnier, K. M., Pinkesz, M., Prince, A. E. R., Liosi, S., Song, L., & Joly, Y. (2019). Potential (mis)use of epigenetic age estimators by private companies and public agencies : Human rights law should provide ethical guidance. Environmental Epigenetics, 5(3). https://doi.org/10.1093/eep/dvz018
- Wang, T., Ma, J., Hogan, A. N., Fong, S., Licon, K., Tsui, B., Kreisberg, J. F., Adams, P. D., Carvunis, A.-R., Bannasch, D. L., Ostrander, E. A., & Ideker, T. (2019). Quantitative translation of dog-to-human aging by conserved remodeling of epigenetic networks (p. 829192). https://doi.org/10.1101/829192
Le grand retour du Lamarckisme ?
Joël de Rosnay parle, déJà en 2011 ici, de « la grande révolution de la biologie de ces cinq dernières années » car elle démontre que dans certains cas, notre comportement agit sur l'expression de nos gènes
Etienne Danchin écrit à ce propos dans un ouvrage sur les formes de transmissions génétique et les autres.
"Lorsqu'au tout début du XIXe siècle Jean-Baptiste de Lamarck a formalisé l'idée que les espèces se transforment au cours du temps, c'était un grand pas dans la connaissance du vivant. L'idée était encore révolutionnaire six décennies plus tard, à l'époque de Darwin. Évidemment, on ne connaissait alors rien des mécanismes d'hérédité, ni de ses règles, ni de la manière dont l'information impliquée était encodée. Lamarck a alors suggéré la «règle de l'usage et du non-usage» des organes comme moteur général de l'évolution. Près de cinquante ans plus tard, Darwin proposait la sélection naturelle.[… selon laquelle] on devrait même s'attendre à l'existence de processus lamarckiens. En effet, l'information génétique, par sa très grande fidélité de transmission, est spécialisée dans la transmission d'informations héritées d'ancêtres très lointains. Or […] l'environnement des ancêtres récents doit constituer un bien meilleur prédicteur de l'environnement dans lequel se développera la génération suivante. Ainsi, tout organisme capable de transmettre à ses descendants des informations sur leur environnement aurait un avantage sélectif majeur, parce que ces derniers seraient en quelque sorte moulés aux conditions environnementales dans lesquelles ils se développeront, survivront et se reproduiront.[…]Tant pis si cette hypothèse est immédiatement taxée de lamarckiste et donc rejetée, car non conforme au néodarwinisme. Il existe dans la littérature scientifique de nombreux faits très bien documentés qui montrent que de tels mécanismes existent bien." Danchin, E. (2021) pp 55-56
- Danchin, E. (2021). L'hérédité comme on ne vous l'a jamais racontée. Humensciences. ici
- Interview dans l'émission Egosystème à la RTS
- Joël de Rosnay, La symphonie du vivant, comment l'épigénétique va changer notre vie (Ed. Les liens qui Libèrent).
Imprimer un modèle 3D d'histone pour le faire manipuler par les élèves ?
Voir à l'écran ou dans un livre ne produit évidemment pas les mêmes apprentissages que la manipulation d'objets tangibles - probablement surtout chez certain.e.s élèves, notamment les plus jeunes ou moins scolaires (Achiam, 2016). On peut par exemple discuter la méthylation en collant un peu de pâte à modeler ou pâte collante genre patafix, blutack sur un modèle en plastique d'une Histone.
Produire de tels modèles est devenu possible, abordable et presque facile… le projet Bioinformatique : opportunités pour l'enseignement fournit les données authentiques ici pour l'histone par exemple afin de la faire imprimer dans un Fablab ou auprès d'un collègue qui a une imprimante 3D.
Fig 9 :Un modèle imprimé en 3D de l'Histone H4 (celui-ci n'a plus qu'un tour d'ADN suite à des manipulations manquant de douceur … )

NB : ne pas oublier de respecter les normes sanitaires ( désinfecter les objets etc)
Sources
La plateforme Jump-To-Science l offre aux membres l'accès a ces articles Comment Obtenir un article mentionné - Achiam, M., Simony, L., & Lindow, B. E. K. (2016). Objects prompt authentic scientific activities among learners in a museum programme. International Journal of Science Education, 38(6), 1012‑1035. https://doi.org/10.1080/09500693.2016.1178869
- Campbell, N. A., & Reece, J. B. (2015). Biology : A global approach (10. ed., global ed). Pearson.
- Childebayeva, A., Goodrich, J. M., Leon-Velarde, F., Rivera-Chira, M., Kiyamu, M., Brutsaert, T. D., Dolinoy, D. C., & Bigham, A. W. (2021). Genome-Wide Epigenetic Signatures of Adaptive Developmental Plasticity in the Andes. Genome Biology and Evolution, 13(2). https://doi.org/10.1093/gbe/evaa239
- Danchin, E. (2021). L'hérédité comme on ne vous l'a jamais racontée. Humensciences.
- Dossin, F., Pinheiro, I., Żylicz, J.J. et al. SPEN integrates transcriptional and epigenetic control of X-inactivation. Nature 578, 455–460 (2020). https://doi.org/10.1038/s41586-020-1974-9
- Dupras, C. (2017). Rapprochement des pôles nature et culture par la recherche en épigénétique : Dissection d'un bouleversement épistémologique attendu. Les ateliers de l'éthique, 12(2‑3), 120. https://doi.org/10.7202/1051278arhttps://doi.org/10.1126/science.aba2340
- Dupras, C., Beck, S., Rothstein, M. A., Berner, A., Saulnier, K. M., Pinkesz, M., Prince, A. E. R., Liosi, S., Song, L., & Joly, Y. (2019). Potential (mis)use of epigenetic age estimators by private companies and public agencies : Human rights law should provide ethical guidance. Environmental Epigenetics, 5(3). https://doi.org/10.1093/eep/dvz018
- Wang, T., Ma, J., Hogan, A. N., Fong, S., Licon, K., Tsui, B., Kreisberg, J. F., Adams, P. D., Carvunis, A.-R., Bannasch, D. L., Ostrander, E. A., & Ideker, T. (2019). Quantitative translation of dog-to-human aging by conserved remodeling of epigenetic networks (p. 829192). https://doi.org/10.1101/829192
- Kayser, J. (2021). Identical twins carry genetic modifications no one else has [Data set]. https://doi.org/10.1126/science.acx9209
- Kreier, Freda (2020) High-altitude living has changed more than just the genes of some Peruvians. https://doi.org/10.1126/science.abg2903
- Li, Z., Zhao, S., Nelakanti, R. V., Lin, K., Wu, T. P., Alderman, M. H., Guo, C., Wang, P., Zhang, M., Min, W., Jiang, Z., Wang, Y., Li, H., & Xiao, A. Z. (2020). N6-methyladenine in DNA antagonizes SATB1 in early development. Nature, 583(7817), 625‑630. https://doi.org/10.1038/s41586-020-2500-9
- McCartney, D. L., Stevenson, A. J., Hillary, R. F., Walker, R. M., Bermingham, M. L., Morris, S. W., Clarke, T.-K., Campbell, A., Murray, A. D., Whalley, H. C., Porteous, D. J., Visscher, P. M., McIntosh, A. M., Evans, K. L., Deary, I. J., & Marioni, R. E. (2018). Epigenetic signatures of starting and stopping smoking. EBioMedicine, 37, 214‑220. https://doi.org/10.1016/j.ebiom.2018.10.051
- Morell, V. (2019). Here's a better way to convert dog years to human years, scientists say. Science. https://doi.org/10.1126/science.aba2340
- Pennisi, E. (2021). Controversial DNA modification could play key role in placenta development. https://doi.org/10.1126/science.abd9820
- Szyf, M. (2014). Lamarck revisited : Epigenetic inheritance of ancestral odor fear conditioning. Nature Neuroscience, 17(1), 2‑4. https://doi.org/10.1038/nn.3603Trotman, J. B., & Calabrese, J. M. (2020). How to silence an X chromosome. Nature, 578(7795), 365‑366. https://doi.org/10.1038/d41586-020-00207-0
- van Dongen, J., Gordon, S. D., McRae, A. F., Odintsova, V. V., Mbarek, H., Breeze, C. E., Sugden, K., Lundgren, S., Castillo-Fernandez, J. E., Hannon, E., Moffitt, T. E., Hagenbeek, F. A., van Beijsterveldt, C. E. M., Jan Hottenga, J., Tsai, P.-C., Min, J. L., Hemani, G., Ehli, E. A., Paul, F., … Boomsma, D. I. (2021). Identical twins carry a persistent epigenetic signature of early genome programming. Nature Communications, 12(1), 5618. https://doi.org/10.1038/s41467-021-25583-
- Wang, T., Ma, J., Hogan, A. N., Fong, S., Licon, K., Tsui, B., Kreisberg, J. F., Adams, P. D., Carvunis, A.-R., Bannasch, D. L., Ostrander, E. A., & Ideker, T. (2019). Quantitative translation of dog-to-human aging by conserved remodeling of epigenetic networks (p. 829192). https://doi.org/10.1101/829192




Aucun commentaire:
Enregistrer un commentaire